 Projekt „The Taverna services for Systems Biology (tav4sb)” jest to usługa internetowa (ang. web service), udostępniająca nowe funkcjonalności z dziedziny biologii systemów rozszerzając w ten sposób możliwości zrębu Taverna Workbench.
Projekt „The Taverna services for Systems Biology (tav4sb)” jest to usługa internetowa (ang. web service), udostępniająca nowe funkcjonalności z dziedziny biologii systemów rozszerzając w ten sposób możliwości zrębu Taverna Workbench.
Taverna Workbench jest narzędziem, które umożliwia projektowanie i wykonywanie eksperymentów in silico. Eksperymenty są tworzone, jako sieci zależności (ang. work-flow), które są przechowywane w postaci plików XML i wykonywane w razie potrzeby, co umożliwia ich łatwe powtarzanie. Sieci zależności składają się z bloków, które są serwisami nazywanymi powszechnie, jako procesory. Techniczne, sieć zależności jest to zbiór procesorów połączonych ze sobą pomiędzy wejściami (ang. inputs), a wyjściami (ang. outputs). Procesory są zaimplementowane, jako web service’y. Taverna umożliwia korzystanie z wielu rozproszonych fizycznie zasobów obliczeniowych i baz danych, a także łączenie ich w zależności.
tav4sb udostępnia następujące funkcjonalności:
- Symulacje numeryczne deterministycznych modeli sieci sygnałowych za pomocą zrębu SBML ODE Solver library (SOSlib).
- Probabilistyczny model-checker formuł Continuous Stochastic Logic (CSL) za pomocą łańcuchów Markowa z czasem ciągłym (CTMC) przy użyciu PRISMa.
- Wizualizacje danych np. trajektorii równań różniczkowych, próbek losowych przy użyciu Mathematici
Serwis korzysta z Symbols Biology Markup Language (SBML) do reprezentacji sieci sygnałowych. Operacje serwisu tav4sb są wykonywane na Gridzie integrującym różne oprogramowania takie, jak Mathematica, PRISM and SBML ODE Solver.
Schemat użycia serwisu serwisu:
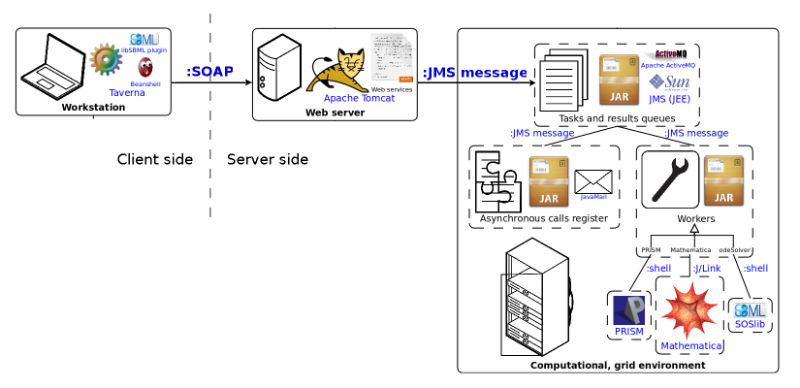
- Użytkownik definiuje oraz uruchamia work-flow zawierający jeden z serwisów tav4sb.
- Taverna łączy się z serwerem za pomocą protokołu SOAP3. Sewer wysyła zadanie do systemu.
- System uruchamia odpowiednie workery i wykonuje zadanie.
- W zależności od serwisu odpowiedź może być synchroniczna, bądź asynchroniczna.
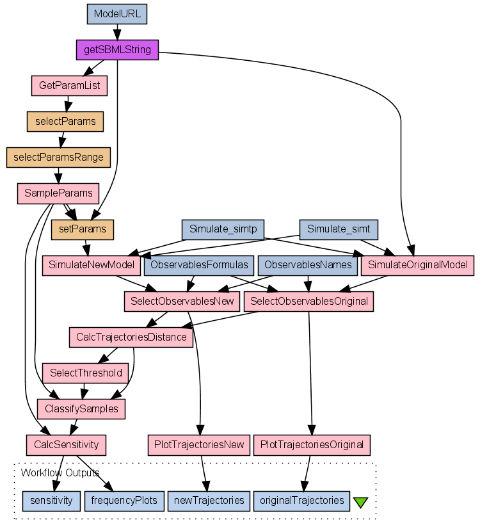
Główną funkcjonalnością tav4sb jest umożliwienie tworzenia eksperymentów wykorzystujących udostępnione metody. Jako przykład możliwości tav4sb może posłużyć wieloparametyrczna analiza wrażliwości modeli biologicznych (ang. multi-parameter sensitivity analysis MPSA) za pomocą metod numerycznych, jak i stochastycznych. MPSA jest to metoda analizy wrażliwości oparta o metodę filtrowania Monte Carlo polegającą na podziale przestrzeni parametrów na regiony akceptowalne i nieakceptowalne.
Stochastyczna wersja metody MPSA skład się z następujących kroków :
- Wybór analizowanych parametrów.
- Ustawienie przedziałów, w których znajdują się parametry.
- Wylosowanie niezależnych próbek.
- Obliczenie błędów próbek.
- Zaklasyfikowanie próbki, jako akceptowalnej, bądź nieakceptowalnej.
- Statystyczne porównanie próbek.
Jednym z wyników metody MPSA są wykresy dystrybuanty empirycznej ( ang. empirical cumulative distribution functions ECDF) próbek akceptowalnych i nieakceptowalnych dla każdego z wybranych parametrów. Ostatecznym rezultatem są dwa rankingi parametrów składające się z wyników porównania dystrybuant empirycznych za pomocą testu Kołmogorowa-Smirnowa oraz współczynnika korelacji Pearsona (ang. Pearson product-moment correlation coeffcient PMCC). Poniżej prezentujemy wyniki stochastycznej wersji MPSA dla prostej reakcji enzymatycznej.
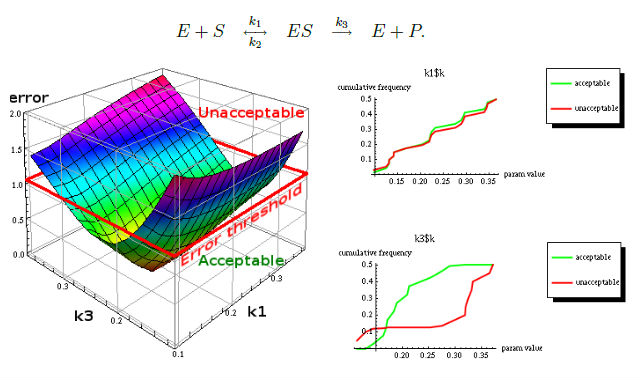
Więcej eksperymentów wraz z dokładnymi opisami jest dostępnych na stronie internetowej projektu http://bioputer.mimuw.edu.pl/tav4sb/?p=ex.
Linki:
Publikacje:
- Rybiński, M. et al., 2012, Tav4SB: integrating tools for analysis of kinetic models of biological systems, BMC Systems Biology, 6(1), p.25.
- Rybiński, M. et al., 2011, Tav4SB: grid environment for analysis of kinetic models of biological systems, ISBRA 2011 Short Abstracts, pp. 92-95.

 Polski
Polski English
English