Bio-Info
W dziale BioInfo portalu znajdują się serwisy dotyczące analizy regulacji ekspresji genów, zwijania łańcuchów RNA i białkowych, przewidywania struktur receptorów, modelowania molekularnego oraz analizy literatury naukowej. Dostępne są wybrane, codziennie uaktualniane bazy danych struktur makrocząsteczek , białek i ich oddziaływań, bazy genomów organizmów pro i eukariotycznych oraz narzędzi i modeli biologii systemów.
Serwisy zgromadzone w tym dziale to m. in.:
- zintegrowany serwis internetowy do przewidywania struktury i funkcji makrocząsteczek biologicznych, białek i RNA;
- serwis internetowy do przewidywania struktur receptorów GPCR i dokowania ligandów oraz ustalenia oddziaływania ligandów na przełączniki molekularne receptora
- narzędzia służące do gromadzenia, zarządzania, analizowania i wizualizowania danych o regulacji ekspresji genów; dane o sekwencji genomowej, genach, obszarach regulatorowych i wystąpieniach miejsc wiążących w skali całych genomów zostaną zintegrowane w bazie danych;
- serwisy bioinformatyczne i medycyny molekularnej do przetwarzania i analizy danych pochodzących z eksperymentów wielkoskalowych: proteomiki, genomiki i transkryptomiki, umożliwiające m.in. automatyczną adnotację białek, tworzenie funkcjonalnych map interakcji między nimi oraz symulacje onkogennych szlaków sygnałowych.
e-LiSe

Program ten znajduję słowa, które często występują razem z podanym przez użytkownika słowem w abstraktach z bazy danych Medline. Umożliwia to użytkownikowi szybką identyfikacje terminów związanych z danym zagadnieniem biologicznym. Jest on szczególnie przydatny w modelowaniu procesu nowotworzenia, bo pomaga szybko ustalić, które geny uczestniczą danym procesie. W przypadku programu E-LiSe użyto analizy statystycznej rozkładu normalnego. Za pomocą parametru Z-score identyfikowano słowa, które.. czytaj dalej »
HT-SAS

Stworzono system automatycznego anotowania białek na podstawie sekwencji homologicznych oraz literatury naukowej. System działa w oparciu o bazę abstraktów literatury naukowej MedLine oraz bazę sekwencji białkowych i anotacji UniProt. Obie bazy zostały zintegrowane przy pomocy bazy danych MySQLa w nowy system w celu szybkiego dostępu do informacji oraz łatwego uaktualniania danych. System umożliwia obecnie automatyczne opisanie dużej liczby sekwencji białkowych przy pomocy słów kluczowych.. czytaj dalej »
MIrOn

Serwis MIRON (Modules Interactions Ontologies) umożliwia znajdowanie funkcjonalnych interakcji pomiędzy wprowadzanymi przez użytkownika sekwencjami białek. Analizuje on publikacje naukowe odnoszące się do poszczególnych sekwencji znajdując wspólne specyficzne słowa kluczowe. Wyniki przedstawiane są w postaci interaktywnych grafów pozwalających ocenić siłę funkcjonalnej interakcji oraz jej składowe. Wizualizacja sieci oddziaływań pozwala użytkownikowi na szybkie stawianie bądź weryfikację hipotez.. czytaj dalej »
Nencki Genomics Database
Baza Danych Genomicznych Instytutu Nenckiego (Nencki Genomics Database – NGD) udostępnia dane o obszarach regulatorowych w skali całych genomów człowieka, myszy i szczura. W przypadku szczura, jest to pierwsza tego typu baza na świecie. Baza NGD jest oparta o referencyjną bazą danych Ensemble funcgen, której zawartość i funkcjonalność poszerza o: możliwość dodawania danych użytkowników, z możliwością zarządzania przez użytkownika dostępem do posiadanych danych, wyniki wyszukiwania.. czytaj dalej »
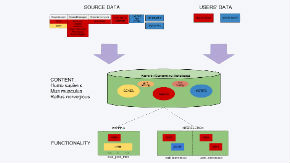
Nencki Genomics Portal
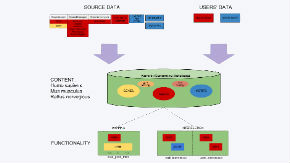
Portal Genomiczny Instytutu Nenckiego (Nencki Genomics Portal – NGP) to grupa powiązanych narzędzi (serwisów internetowych) i baz danych służących do analizy regulacji transkrypcyjnej. Funkcjonalność portalu jest dostępna z poziomu przeglądarki internetowej, za pośrednictwem naszego serwera Galaxy. Dostępne za pośrednictwem portalu aktywności są pogrupowane są w 4 wzajemnie powiązane grupy: analiza genomu; analiza ekspresji; analiza regulacji; analiza funkcji. W obrębie każdej grupy dostępnych.. czytaj dalej »
Polskie lustro wybranych baz danych biologicznych

Infrastruktura informatyczna IBB PAN została specjalnie przystosowana do kooperacji z największym ośrodkiem bioinformatycznym Europy – European Bioinformatics Institute (EBI). W ramach tej współpracy IBB PAN stał się w pełni funkcjonalnym lustrem serwisów bioinformatycznych i biomedycznych udostępnianych przez EBI. W ramach pierwszego etapu prac zostały udostępnione następujące bazy danych oraz towarzyszące im serwisy: UniProt Knowledge Database (UniProtKB) Molecular Structure Database.. czytaj dalej »
Serwisy do modelowania struktury i funkcji makrocząsteczek
Samodzielne serwisy: QA-RecombineIt: a server for quality assessment and recombination of protein models http://genesilico.pl/qarecombineit Metapredictor of protein-RNA interactions (integrated with GeneSilico metaserver) http://iimcb.genesilico.pl/meta2/ MetaDisorder: a meta-server for the prediction of intrinsic disorder in proteins http://genesilico.pl/metadisorder/ AmIGoMR: a server for testing the utility of comparative models and their local quality for protein crystal structure.. czytaj dalej »
Zintegrowany serwis do modelowania struktur receptorów GPCR i dokowania ligandów
GPCRM – serwis do modelowania przez homologię struktur receptorów GPCR. GPCRM-DOCK – serwis do dokowania ligandów do receptorów GPCR. czytaj dalej »

 Polski
Polski English
English