Bio-Info
W dziale BioInfo portalu znajdują się serwisy dotyczące analizy regulacji ekspresji genów, zwijania łańcuchów RNA i białkowych, przewidywania struktur receptorów, modelowania molekularnego oraz analizy literatury naukowej. Dostępne są wybrane, codziennie uaktualniane bazy danych struktur makrocząsteczek , białek i ich oddziaływań, bazy genomów organizmów pro i eukariotycznych oraz narzędzi i modeli biologii systemów.
Serwisy zgromadzone w tym dziale to m. in.:
- zintegrowany serwis internetowy do przewidywania struktury i funkcji makrocząsteczek biologicznych, białek i RNA;
- serwis internetowy do przewidywania struktur receptorów GPCR i dokowania ligandów oraz ustalenia oddziaływania ligandów na przełączniki molekularne receptora
- narzędzia służące do gromadzenia, zarządzania, analizowania i wizualizowania danych o regulacji ekspresji genów; dane o sekwencji genomowej, genach, obszarach regulatorowych i wystąpieniach miejsc wiążących w skali całych genomów zostaną zintegrowane w bazie danych;
- serwisy bioinformatyczne i medycyny molekularnej do przetwarzania i analizy danych pochodzących z eksperymentów wielkoskalowych: proteomiki, genomiki i transkryptomiki, umożliwiające m.in. automatyczną adnotację białek, tworzenie funkcjonalnych map interakcji między nimi oraz symulacje onkogennych szlaków sygnałowych.
ClustSense
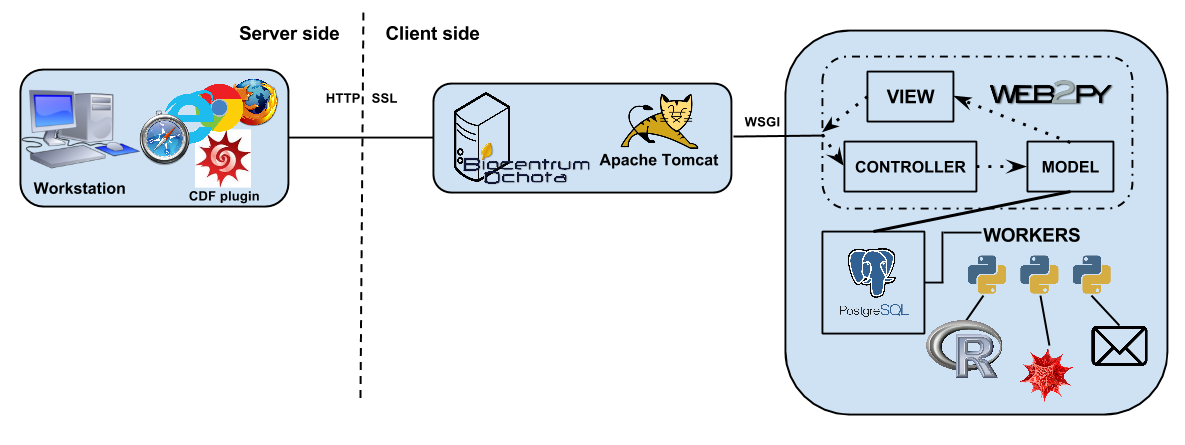
ClustSense: web-serwis do symulowania i analizy modeli (onkogennych) szlaków sygnałowych. Funkcjonalność: Symulacje numeryczne i wizualizacje. Analiza wrażliwości. Klastrowanie parametrów i analiza identyfikowalności. Workflow Architektura Metody Przykład użycia Publikacje : Michał Włodarczyk, Karol.. czytaj dalej »
DEDAL
DEDAL is a protein structure alignment tool which is independent of rigid-body constraints. This is carried out by systematically evaluating local structure context to identify similar regions of proteins. By focusing on local structure and performing a spatial rather than sequence attached analysis of matching substructures it is not constrained by any particular order of structural features along the protein sequence. Because it identifies all local similarities between compared structures, it.. czytaj dalej »
DEDAL
DEDAL (DEscriptor Defined ALignment) służy do obliczania uliniowień strukturalnych dla podanej pary struktur białek bez traktowania ich jako obiekty sztywne. Uliniowienie obliczane jest na podstawie lokalnych podobieństw, z których następnie składane jest globalne uliniowienie. Dzięki definicji lokalnego deskryptora, który obejmuje przestrzenne (nie sekwencyjne) otoczenie wybranej reszty aminokwasowej, możliwe jest odstąpienie od wymuszania jednakowego nanizania podobnych fragmentów na sekwencje.. czytaj dalej »
DiSCuS
DiSCuS – Database System for Compound Selection Głównym celem systemu DiSCuS jest wspomaganie naukowców w analizie danych pochodzących z wielko skalowego wirtualnego screeningu (HTVS), a w szczególności wyselekcjonowaniu związku wiodącego do dalszych badań, syntezy czy zakupu. Aby osiągnąć powyższe DiSCuS posida następujące właściwości: miejsce do organizacji kompleksów ligand-receptor (pochodzących z niezależnych źródeł), narzędzia do oceny jakości tych kompleksów, np. funkcje oceny.. czytaj dalej »
Essentia Proteomica
The Essential Proteomica portal employs a WeBIAS server developed in MMRC. It is used to publish bioinformatics services. It handles query submissions with XML defined forms, input data verification, job queueing and presentation of results. Users may create accounts to gain access to archival results and other advanced functions. The following services are presently available on the Essentia Proteomica portal: DEDAL – structural alignments of proteins ResiCon – computation of dynamic domains in.. czytaj dalej »
Essentia Proteomica
Portal Essentia Proteomica jest oparty na stworzonym w IMDiK oprogramowaniu WeBIAS i służy do publikowania usług bioinformatycznych. Obsługuje on przyjmowanie zapytań za pośrednictwem łatwo definiowalnych formularzy, weryfikację poprawności danych wejściowych, kolejkowanie zadań i wizualizację wyników. Użytkownicy mogą zakładać konta na portalu w celu uzyskania dostępu do archiwalnych wyników obliczeń i innych zaawansowanych funkcji. Obecnie w ramach portalu Essentia Proteomica udostępniane są.. czytaj dalej »
ResiCon
ResiCon is a method of finding dynamic domains (quasi-rigid structural parts) in proteins by analyzing an ensemble of structures, provided in a PDB file. ResiCon uses a time-generalized concept of contacts between amino acids to construct a virtual scaffold representing protein’s rigidity (see [1] for the description of local structural properties, including contacs). Next, ResiCon performs a spectral clustering, identifying quasi-static regions as clusters.. czytaj dalej »
ResiCon
ResiCon to metoda wyznaczająca dynamiczne domeny (pół-sztywne elementy strukturalne) w białkach, poprzez analizę zbioru struktur zawartych w pliku PDB. ResiCon konstruuje wirtualne rusztowanie zawieszone na aminokwasach danego białka, w którym łączenia reprezentują sztywność kontaktów między-aminokwasowych. Użyta w tym celu koncepcja kontaktu jest uogólnieniem wcześniejszego pomysłu, wykorzystanego w [1] do opisu deskryptorów lokalnej struktury. Następnie, ResiCon przeprowadza klastering.. czytaj dalej »
TavOPS

Projekt „The Taverna services for Systems Biology (tav4sb)” jest to usługa internetowa (ang. web service), udostępniająca nowe funkcjonalności z dziedziny biologii systemów rozszerzając w ten sposób możliwości zrębu Taverna Workbench. Taverna Workbench jest narzędziem, które umożliwia projektowanie i wykonywanie eksperymentów in silico. Eksperymenty są tworzone, jako sieci zależności (ang. work-flow), które są przechowywane w postaci plików XML i wykonywane w razie potrzeby, co.. czytaj dalej »
Tools4miRs
Tools4miRs to baza publikacji dotyczących narzędzi umożliwiających szerokopojętą analizę cząsteczek miRNA. Obecnie zebrane publikacje opisują algorytmy/metody do przewidywania prekursorów miRNA, przewidywania targetów miRNA, przewidywania nowych miRNA, analizy danych otrzymanych z sekwencjonowania małych RNA oraz różnorodne bazy danych. W przyszłości znajdzie się tutaj również serwis skupiający wszystkie te narzędzia.” http://bco.ibb.waw.pl/wp-content/plugins/zotpress/ nature 2440986 [1]M.. czytaj dalej »

 Polski
Polski English
English