Edukacja
Dział Edukacja portalu zawiera wybrane serwisy skierowane m.in. do studentów pierwszego, drugiego i trzeciego stopnia (MU). Obejmują one głównie wizualizację procesów biologicznych oraz umożliwiają przeglądanie wiedzy zgromadzonej w bazach danych konsorcjum Biocentrum Ochota. Serwisy te mogą być wykorzystane do szkolenia studentów w zakresie zastosowania technik informatycznych w biologii, medycynie i przemyśle.
Poniżej znajduje się lista serwisów oraz opis ich wykorzystania
3d Brain Atlas Reconstructor

Zestaw narzędzi do konstrukcji trójwymiarowych atlasów mózgów na podstawie dwuwymiarowych danych oraz usługę internetowa oraz zbiór ogólnodostępnych atlasów różnych gatunków zwierząt. Linki: http://service.3dbar.org http://3dbar.org Referencje: Piotr Majka, Ewa Kublik, Grzegorz Furga, Daniel K. Wójcik. 2012. Common Atlas Format and 3D Brain Atlas Reconstructor, the infrastructure for constructing 3D brain atlases. Neuroinformatics, 10:181-197 (doi:10.1007/s12021-011-9138-6) Piotr Majka, Jakub.. czytaj dalej »
dbBCO – Baza publikacji konsorcium
Baza publikacji konsorcium Biocentrum Ochota. Baza skupia wszystkie publikacje naukowe w których wykorzystana była infrastruktura informatyczna zbudowana w ramach konsorcium. Publikacje posiadają odpowiednie odnośniki, style oraz możliwość załadowania pełnej wersji tekstu tam gdzie licencja wydawcy na to pozwala. http://bco.ibb.waw.pl/wp-content/plugins/zotpress/ nature 338254 1.Kaczanowski, S., Siedlecki, P. & Zielenkiewicz, P. The High Throughput Sequence Annotation Service (HT-SAS).. czytaj dalej »
e-LiSe

Program ten znajduję słowa, które często występują razem z podanym przez użytkownika słowem w abstraktach z bazy danych Medline. Umożliwia to użytkownikowi szybką identyfikacje terminów związanych z danym zagadnieniem biologicznym. Jest on szczególnie przydatny w modelowaniu procesu nowotworzenia, bo pomaga szybko ustalić, które geny uczestniczą danym procesie. W przypadku programu E-LiSe użyto analizy statystycznej rozkładu normalnego. Za pomocą parametru Z-score identyfikowano słowa, które.. czytaj dalej »
Tools4miRs
Tools4miRs to baza publikacji dotyczących narzędzi umożliwiających szerokopojętą analizę cząsteczek miRNA. Obecnie zebrane publikacje opisują algorytmy/metody do przewidywania prekursorów miRNA, przewidywania targetów miRNA, przewidywania nowych miRNA, analizy danych otrzymanych z sekwencjonowania małych RNA oraz różnorodne bazy danych. W przyszłości znajdzie się tutaj również serwis skupiający wszystkie te narzędzia.” http://bco.ibb.waw.pl/wp-content/plugins/zotpress/ nature 2440986 [1]M.. czytaj dalej »
VirRespir
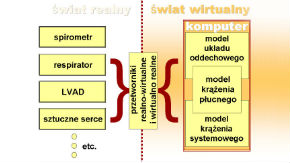
Idea projektu Istnieje szereg modeli układu oddechowego (UO), ale zazwyczaj co najmniej jeden rodzaj zjawisk fizjologicznych albo jest brany pod uwagę w dużym uproszczeniu albo w ogóle jest ignorowany. Na przykład następujące aspekty są często pomijane: wpływ grawitacji i innych czynników na rozkład wentylacji i perfuzji, taki opis matematyczny oskrzeli, który umożliwia poprawną symulację przepływu powietrza podczas zarówno spokojnych wdechów i wydechów jak i forsownych (jak podczas spirometrii).. czytaj dalej »

 Polski
Polski English
English